Responsabile: Prof. Gianni Barcaccia
Collaboratori: Dott. Alessandro Vannozzi e Dott. Fabio Palumbo
Lista dei servizi proposti dal nostro laboratorio:
Marcatori del DNA per l’analisi genica, genetica e genomica in specie di interesse agro-alimentare (RFLP e PCR-derivati, inclusi SSR e SNP)
• DNA fingerprinting
• DNA genotyping
• DNA haplotyping
• cp/mtDNA barcoding
• MG/QTL mapping
• MAS breeding
• mRNA profiling
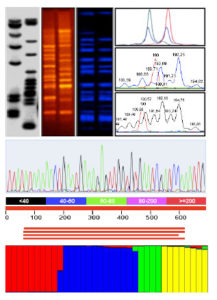
Tecnologia che consente la discriminazione tra individui nonché l’identificazione univoca di singoli individui di una data specie in base ai profili elettroforetici di marcatori molecolari dominanti (AFLP) rilevati nel loro genoma mediante ibridazione o amplificazione.
Tecnologia che consente la determinazione del genotipo di uno o più individui mediante l’analisi di marcatori molecolari codominanti (SSR): la stima del grado di omozigosi/eterozigosi è basata sulla composizione allelica ad un certo numero di loci mappati (>10).
Tecnologia che consente la definizione della composizione nucleotidica delle possibili varianti alleliche con riferimento ad una singola sequenza genica: la ricostruzione dell’aplotipo è basata sulla ricerca di polimorfismi per singoli nucleotidi (SNP).
Tecnologia che consente l’identificazione genetica di un individuo usando una breve sequenza nucleotidica di un gene bersaglio (BARCODE) di origine plastidiale o mitocondriale attraverso la comparazione con una banca dati di sequenze di geni omologhi di specie tassonomicamente note.
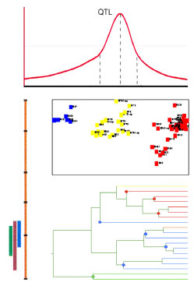
Analisi genetico-molecolare di popolazioni segreganti per il mappaggio di geni Mendeliani e QTL; Identificazione di sequenze geniche e caratterizzazione dei loro modelli di espressione.
Analisi elettroforetica e spettrofotometrica di DNA genomico e di RNA totale.
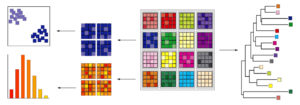
Caratterizzazione bioinformatica e annotazione ontologica (GO) di dataset di sequenze genomiche (DNA) e trascrittomiche (EST) e di polimorfismi genici (marcatori molecolari).
Determinazione di statistiche di diversità genetica, similarità genetica, identità genetica, differenziazione genetica e flusso genico, distanze genetiche, gradi di omozigosi/eterozigosi, indice di fissazione (coefficienti di inbreeding, F) e predizione di eterosi (heterosis, H).
Caratterizzazione genomica di linee pure, ibridi, cloni, sintetiche, ecotipi, ecc. con marcatori molecolari dominanti e co-dominanti.
Determinazione della diversità genetica a livello intra- ed inter-specifico usando marcatori molecolari di origine nucleare, mitocondriale e plastidiale.
Identificazione genetica di prodotti alimentari a partire da qualsiasi matrice solida e liquida utile ai fini della tracciabilità lungo la filiera.
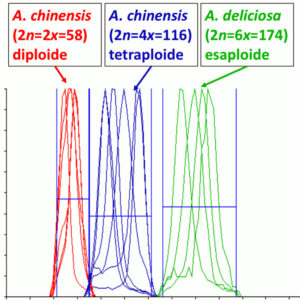
Determinazione del livello di ploidia (2x, 3x, 4x, ecc.) e della dimensione del genoma (Mpb) di piante mediante citometria a flusso (Flow cytometry, FCM)